上一篇文章讨论了如何求一个数据集的前n个主成分。我们虽然求出了这些主成分所代表的坐标轴的方向,但数据集本身依然是n维的,并没有进行降维。具体我们如何运用PCA对数据进行映射的呢?本篇文章将探讨高维数据向低位数据进行映射的过程。
一、把高维数据映射为低位数据的思路
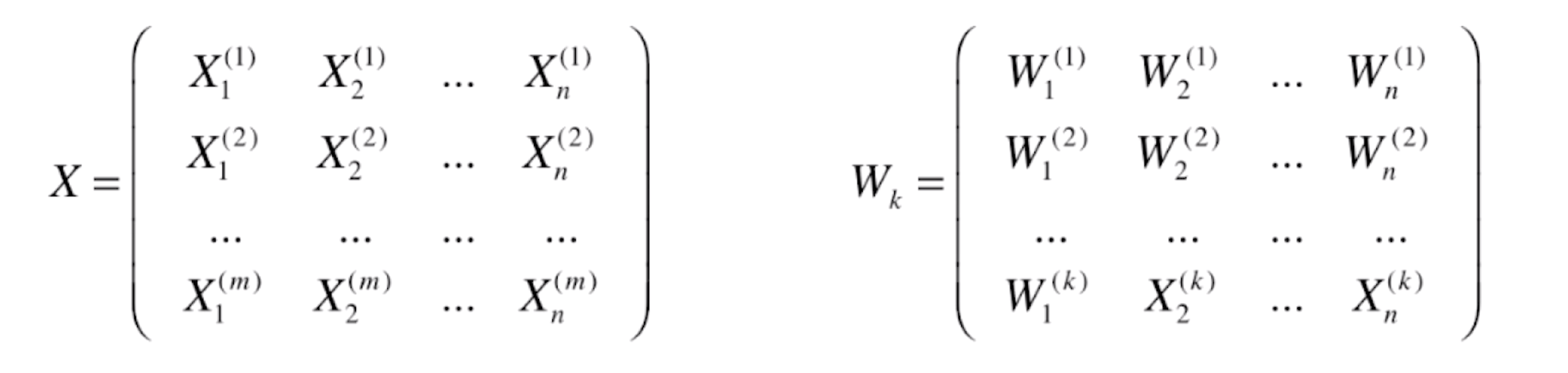
如上图所示,矩阵 X 有 m 行 n 列,代表 m 个样本 n 个特征。假设通过主成分分析法,我们已经求出了针对数据集 X 来说的前 k 个主成分,记作 Wk 。它一共有 k 行,每一行代表每一个主成分的单位方向,Wk 的列数一共有 n 列,代表每一个主成分的坐标轴应该是有 n 个元素。
主成分分析的本质是从一个坐标系转换到另外一个坐标系,若原来的坐标系有 n个维度,转换后的坐标系也有 n个维度。只不过对于转换后的坐标系,我们只取出前 k个,因为这 k个方向更加重要(更能描述数据集的特征),于是就形成了 Wk 这个 k*n的矩阵。
那我们如何将 X 从 n 维转换成 k 维呢?前面我们提到,我们使用一个样本和一行 W 进行点乘,点乘的结果就是将这个样本映射到 W 这个轴上得到的模长:
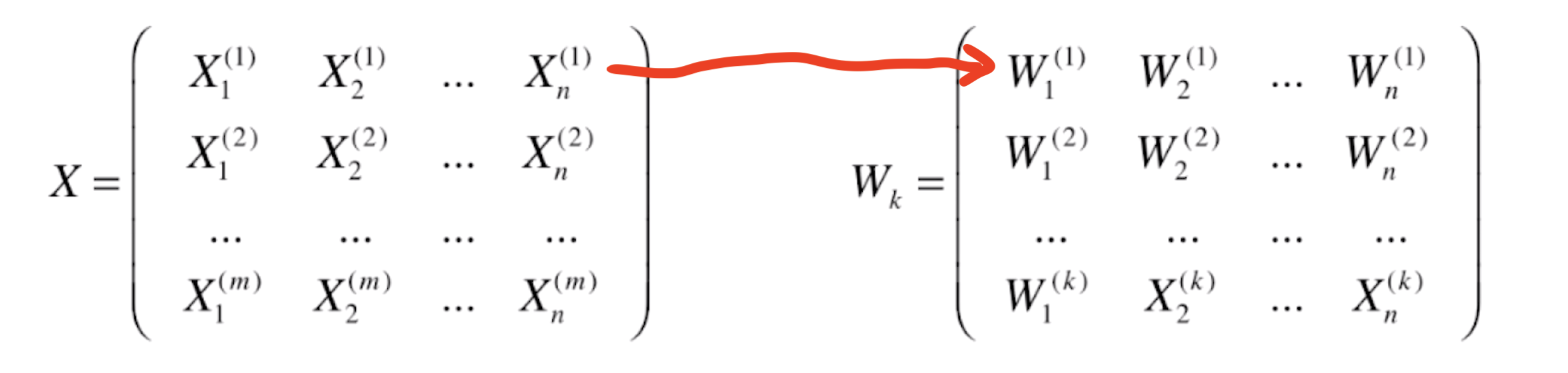
相应的,如果我们将一个样本和 k 个 W(每一行)分别进行点乘,那就得到了这一个样本在这 k 个方向上相应的每一个方向上的大小(模长):
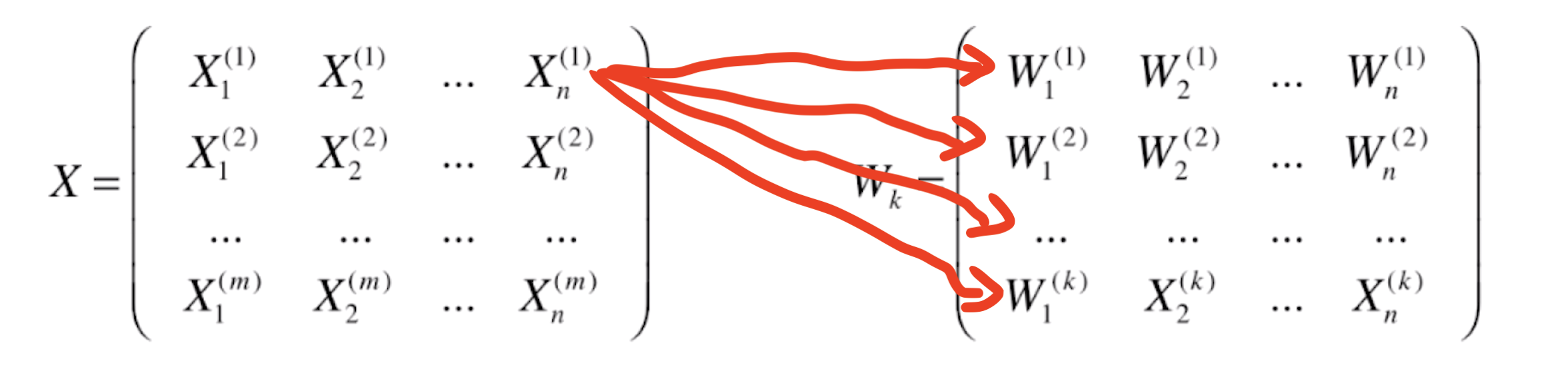
那么这 k 个元素合在一起,就能表示我们一个样本映射到新的 k 个轴所对应的坐标系上相应的样本的大小。简而言之,我们的样本1(X(1) )依次去乘以 W(1) 、W(2) 、W(3) ……一直到W(k),这 k 个数组成的向量,是 X(1) 映射到 Wk 这个坐标系上得到的一个新的 k 维向量。由于 k < n,我们就完成了一个样本从 n 维到 k 维的映射。这个过程以此类推,我们对X(2) 、X(3) 、……X(m) 都这样做,我们就将所有的样本从 n 维映射到了 k 维。
其实,我们整体就做了这样的一个矩阵乘法: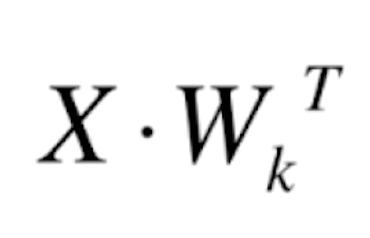 ,根据矩阵乘法的性质,我们把这个矩阵乘法的结果记作 Xk,那么就有:
,根据矩阵乘法的性质,我们把这个矩阵乘法的结果记作 Xk,那么就有:
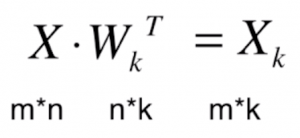
其中 Xk 是一个 m*k 的矩阵。这样我们就完成了高维数据到低位数据的映射。
当然,我们成功求出了一个 m*k 的矩阵 Xk ,反过来,我们也可以通过 Wk 把它恢复成 m*n 的矩阵。
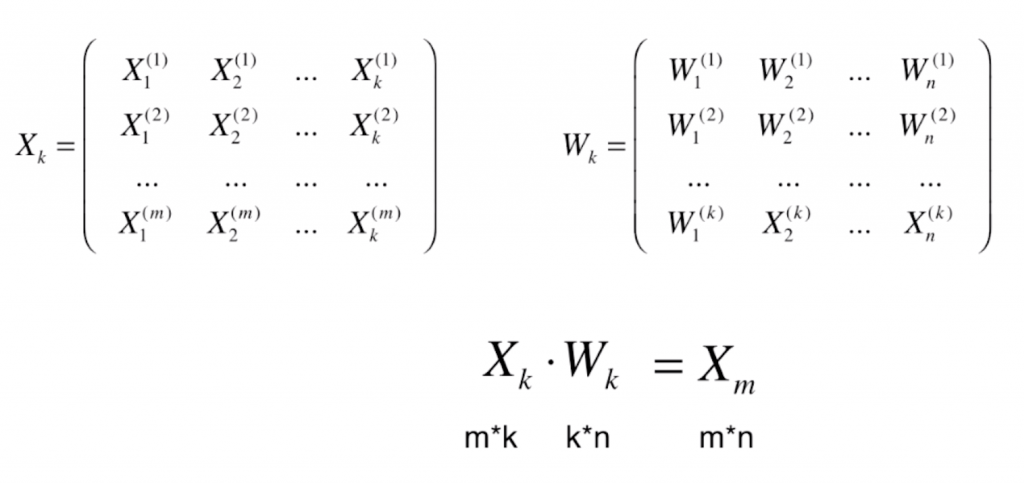
二、编程实现映射和恢复
在MyML包的PCA.py文件的PCA类的最后新增下面transform和inverse_transform两个函数即可:
import numpy as np
class PCA:
def __init__(self, n_components):
#初始化PCA
assert n_components >= 1, "n_components must be valid"
self.n_components = n_components
self.components_ = None
def fit(self, X, eta=0.01, n_iters=1e4):
#获得数据集X的前n个主成分
assert self.n_components <= X.shape[1], \
"n_components must not be greater than the feature number of X"
def demean(X):
return X - np.mean(X, axis=0)
def f(w, X):
return np.sum((X.dot(w) ** 2)) / len(X)
def df(w, X):
return X.T.dot(X.dot(w)) * 2. / len(X)
def direction(w):
return w / np.linalg.norm(w)
def first_component(X, initial_w, eta=0.01, n_iters=1e4, epsilon=1e-8):
w = direction(initial_w)
cur_iter = 0
while cur_iter < n_iters:
gradient = df(w, X)
last_w = w
w = w + eta * gradient
w = direction(w)
if (abs(f(w, X) - f(last_w, X)) < epsilon):
break
cur_iter += 1
return w
X_pca = demean(X)
self.components_ = np.empty(shape=(self.n_components, X.shape[1]))
for i in range(self.n_components):
initial_w = np.random.random(X_pca.shape[1])
w = first_component(X_pca, initial_w, eta, n_iters)
self.components_[i,:] = w
X_pca = X_pca - X_pca.dot(w).reshape(-1, 1) * w
return self
def transform(self, X):
#将给定的X,映射到各个主成分分量中
assert X.shape[1] == self.components_.shape[1]
return X.dot(self.components_.T)
def inverse_transform(self, X):
#将给定的X,反向映射回原来的特征空间
assert X.shape[1] == self.components_.shape[0]
return X.dot(self.components_)
def __repr__(self):
return "PCA(n_components=%d)" % self.n_components
三、PCA类的实际使用
在工程的main.py中,实现以下代码,来使用我们上面定义好的PCA类:
import numpy as np
import matplotlib.pyplot as plt
X = np.empty((100, 2))
X[:,0] = np.random.uniform(0., 100., size=100)
X[:,1] = 0.75 * X[:,0] + 3. + np.random.normal(0, 10., size=100)
from MyML.PCA import PCA
pca = PCA(n_components=2)
pca.fit(X)
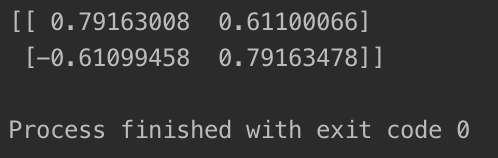
print(pca.components_) #打印两个主成分的方向
pca = PCA(n_components=1)
pca.fit(X)
X_reduction = pca.transform(X) #对X进行降维
X_restore = pca.inverse_transform(X_reduction) #恢复降维后的X
#绘制X和X_restore
plt.scatter(X[:,0], X[:,1], color='b', alpha=0.5)
plt.scatter(X_restore[:,0], X_restore[:,1], color='r', alpha=0.5)
plt.show()
打印的主成分方向和绘制的图像如下:
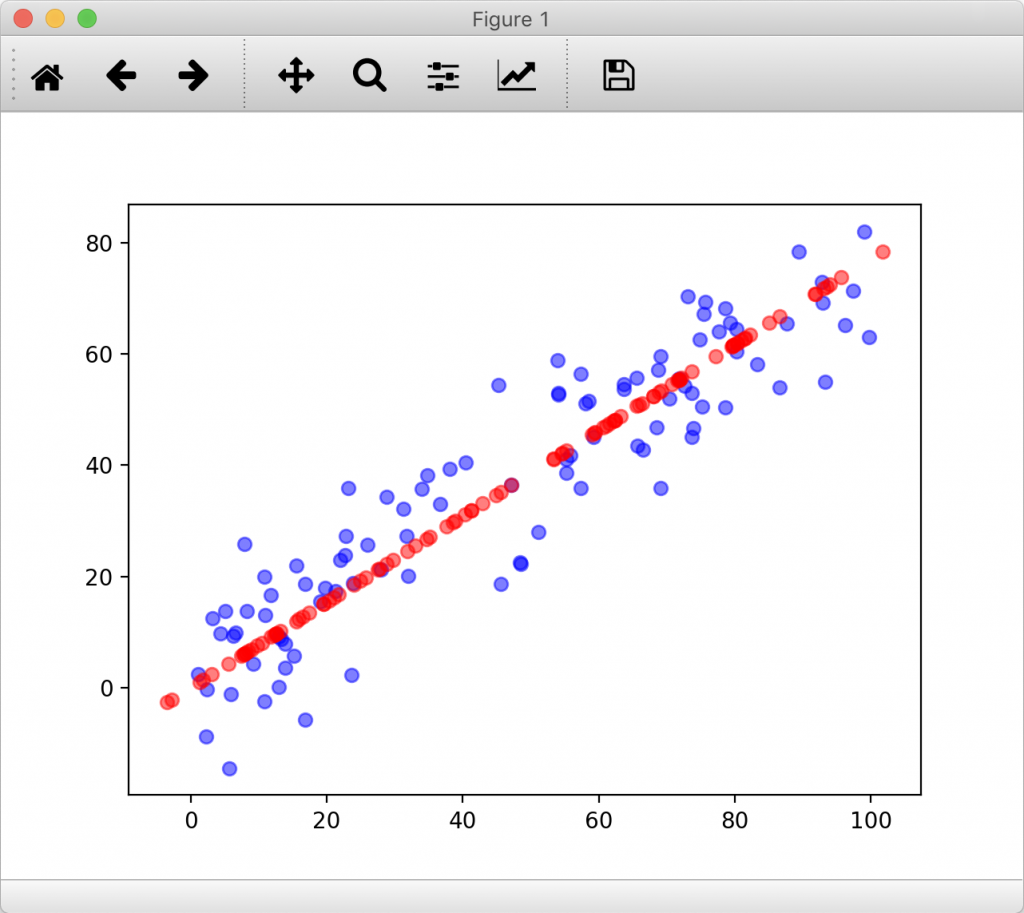
在降维的过程中会丢失信息,restore的过程并不能恢复信息,restore只不过是在高维的空间里表达低维的样本而已,就像上图这些红色的点。
最后我们再来梳理一遍PCA降维的基本原理:首先PCA做的事是寻找另外一个坐标系,这个坐标系中的每一个轴依次可以表达原来样本的重要程度,也就是所谓的“主成分”。取出前 k 个最重要的主成分,然后就可以将所有的样本映射到这 k 个轴上,得到一个低维的数据信息。